学习北京大学《生物信息学-导论与方法》-高歌、魏丽萍老师课程的笔记梳理,喜欢本课程推荐去Coursera观看学习袄~
课程材料采用 CC BY-NC-SA 协议开放共享。
大部分是整理的两位老师说的话,其中的“我”多指两位老师,根据语境判断,不要太较真哈
大纲
1.1 课程介绍
1.2 什么是生物信息学?
1.3 生物信息学历史
1.4 中国大陆的生物信息学
1.5 阅读参考
1.6 测试题
1.7 参考答案
1.1 课程介绍
- Needleman-Wunsch 全局比对算法:1970
- PAM矩阵:1978,两个氨基酸比对在一起的的优劣程度的评估
- Smith-Waterman 局部比对算法:1981
- Gapped-BLAST 算法:1990
- BLOSUM 矩阵:1992
- PSI-BLAST 算法:1997
- Gene Ontology、KEGG 数据库
- 马尔可夫模型及隐马尔可夫模型
- 什么是马尔可夫模型和隐马尔可夫模型
- 如何构建这些模型、如何对模型进行训练、怎样用这些模型进行预测
- 如何通过马尔可夫模型进行序列比对
- 如何用隐马尔可夫模型预测给定DNA序列中的编码区域
- 这些模型如何扩展应用于对基因的预测
- 基因预测工具GeneScan
- 生物信息学重要的数据库和软件资源
- 着重于比较大的常用资源
- 遗传变异呢SNP、SNV
- MAQ
- BWA
- GATK
- 如何利用已有知识建立预测模型的统计问题
- SIFT
- PolyPhen
- SAPRED
- 基因表达
- 基因差异表达
- 计算基因表达量
- 计算表达差异的统计显著性
- 可变剪切体
- 如何把短片段拼接成剪接体
- Cufflinks,2010
- SVAP,高歌、孔雷
- 转录本编码
- CPC 软件,2007
- 判断转录本是否编码蛋白
- 非编码RNA可能的功能
- 利用已知的编码和非编码RNA,建立RNA编码潜能预测模型,找到非编码RNA的特征
- 案例研究1:新基因的起源与演化
- 案例研究2:DNA甲基化酶的功能演化
1.2 什么是生物信息学?
知识点:
- 每个正常的人的体细胞有多少条染色体?
- 23对46条
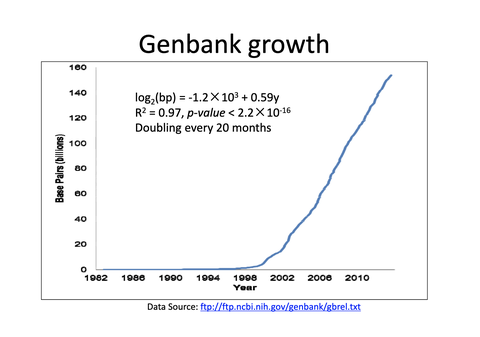
- Human genome has 3.1 billion(3.1 * 10^9^) base pairs
- ~ 2.9% of the bases encode genes
- 一个基因可能有多个剪切体,翻译成不同蛋白
- Drosophila/果蝇的DSCAM1基因,一个基因有38000多个可变剪切体
- ~ 97% of the genome were previously called "junk"
- 但现在我们知道这些区间其实包含了大量的
- regulatory/调控元素, 它决定着
- 在哪里、什么时间、表达哪些蛋白, 表达多少
- 世界上除了RNA病毒(AUCG)之外其他的所有物种的基因组都是由ATCG这样简单的重复组成
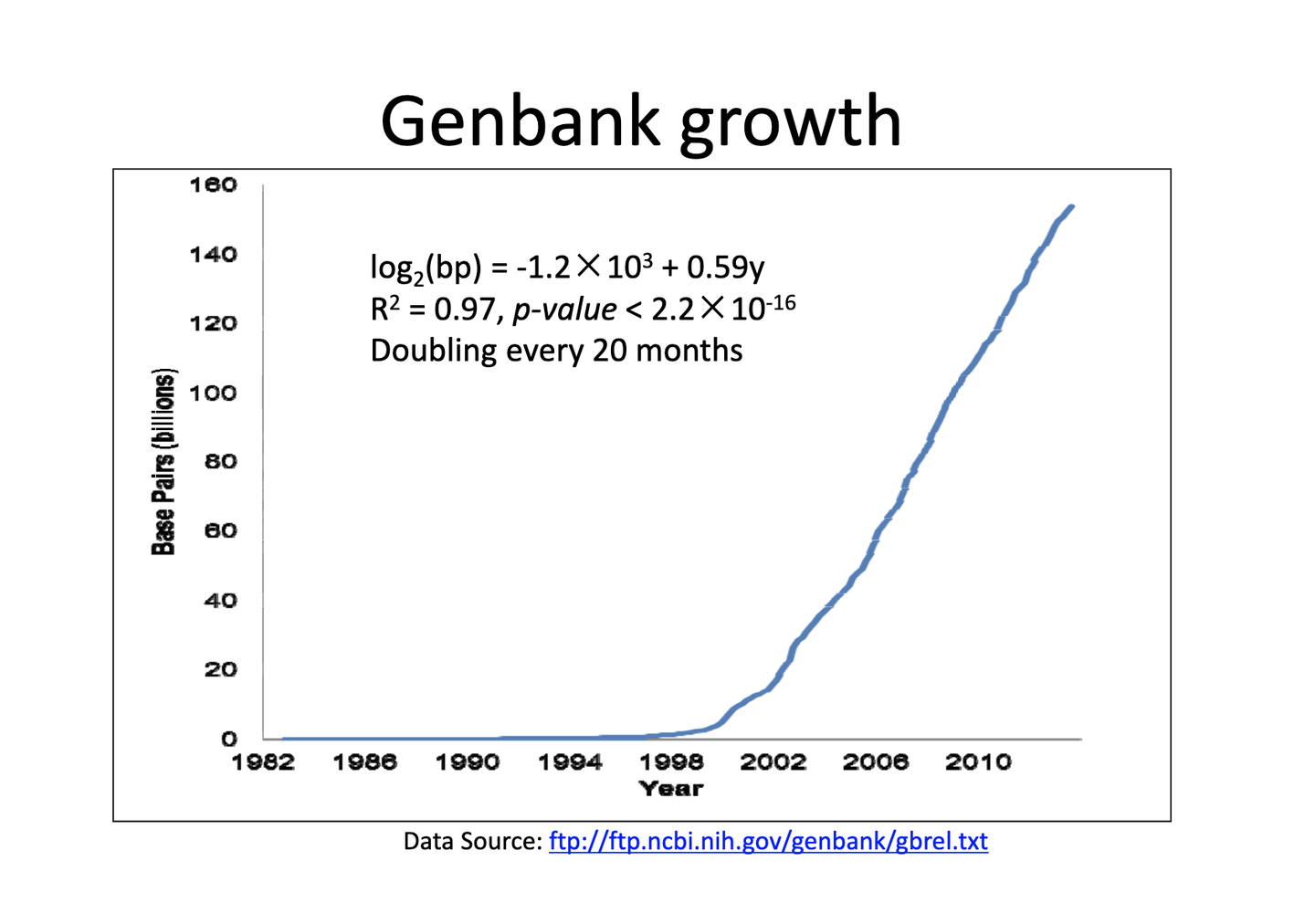 image-20200804234859951
image-20200804234859951
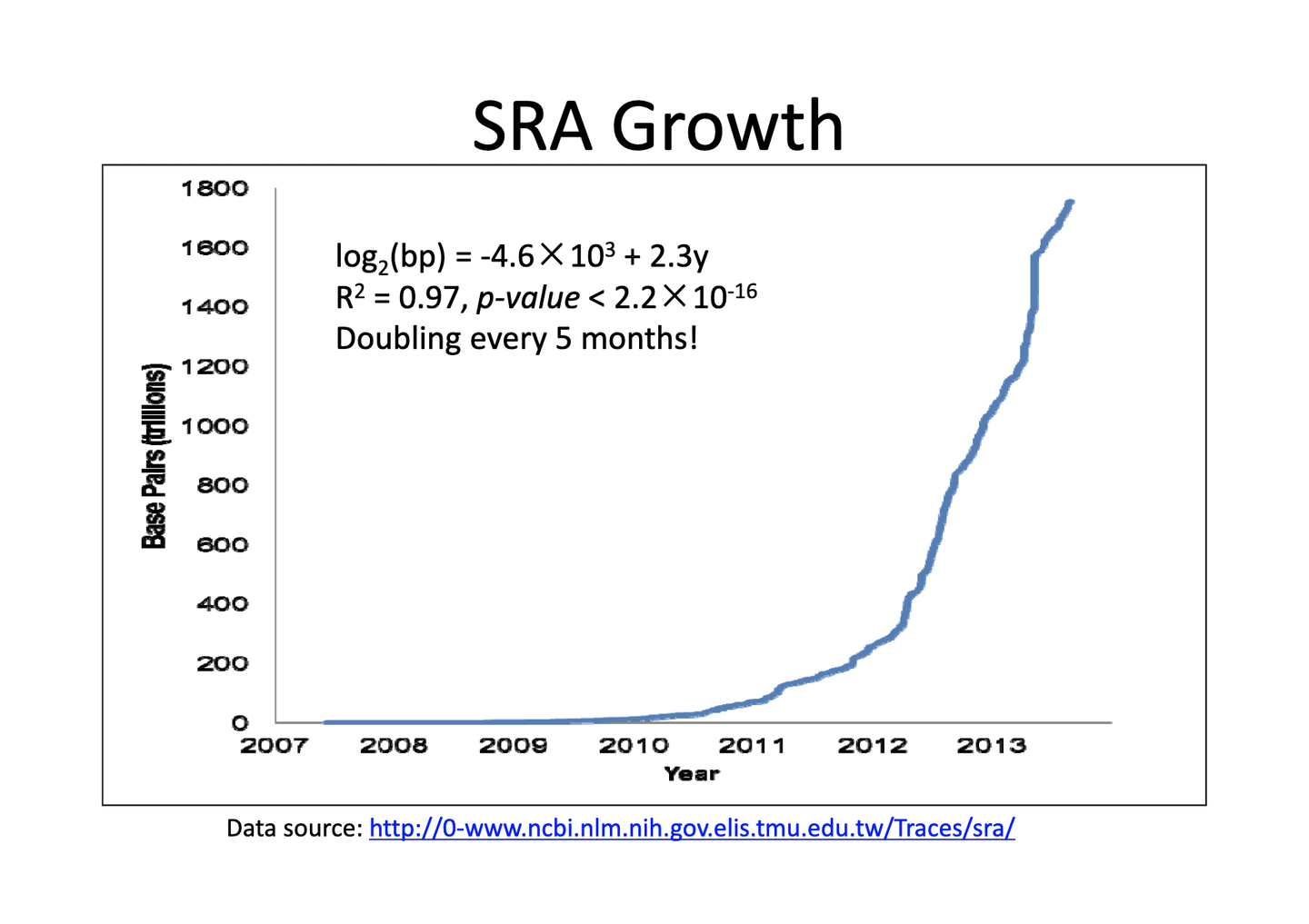 image-20200804235111628
image-20200804235111628
- 这给生命科学带来了巨大的机遇,21世纪是生物的世纪!
 image-20200804235001058
image-20200804235001058
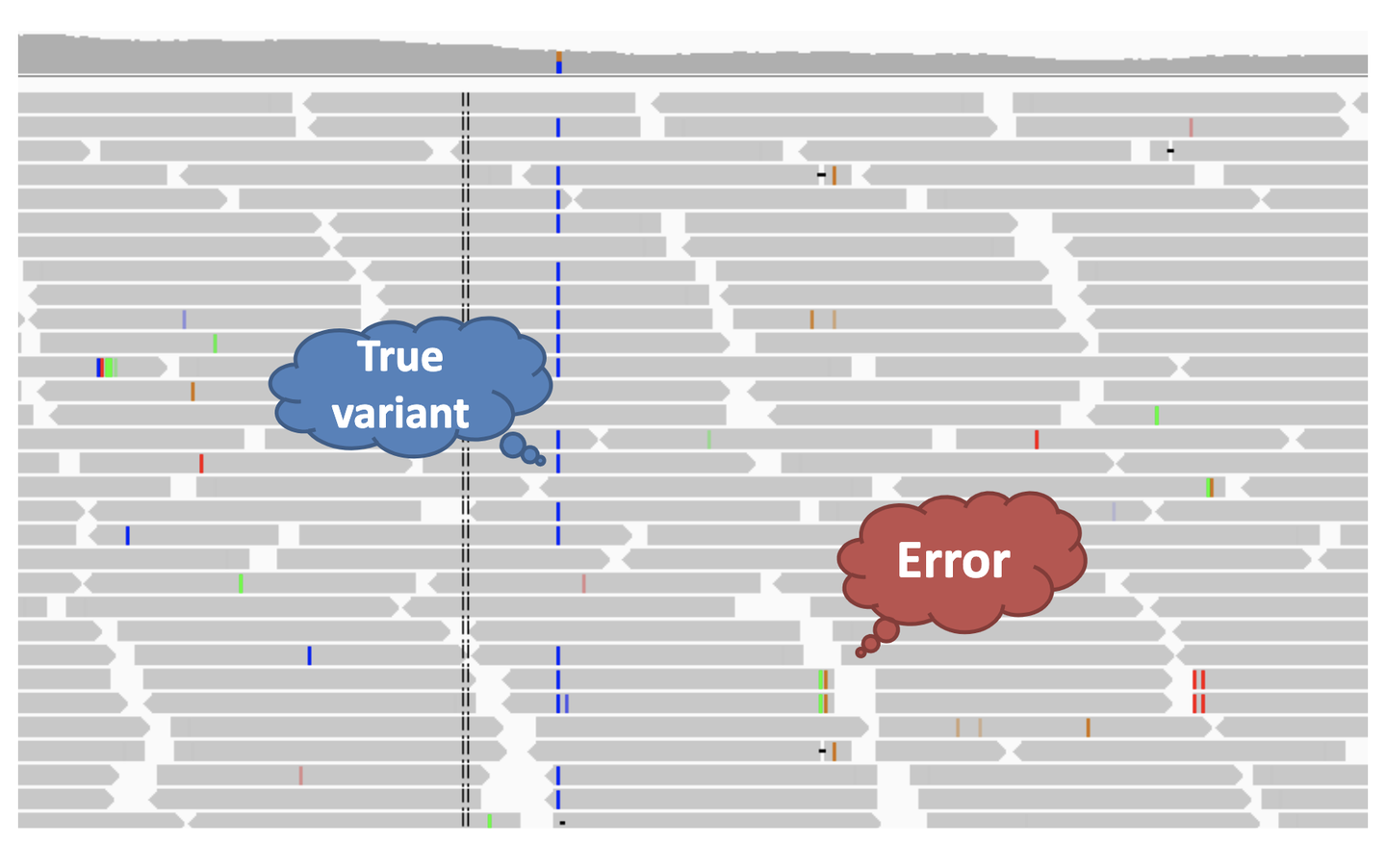 image-20200804235235784
image-20200804235235784
- 二代测序技术单个碱基、单次测序的错误率是传统Sanger测序错误率的100倍高
- 如何从这样大量的错误里找到真正的信号? 这在技术上是有很大的挑战的。
- 所以在有这样的技术挑战的时候,其实就是一 个技术创新的机遇。
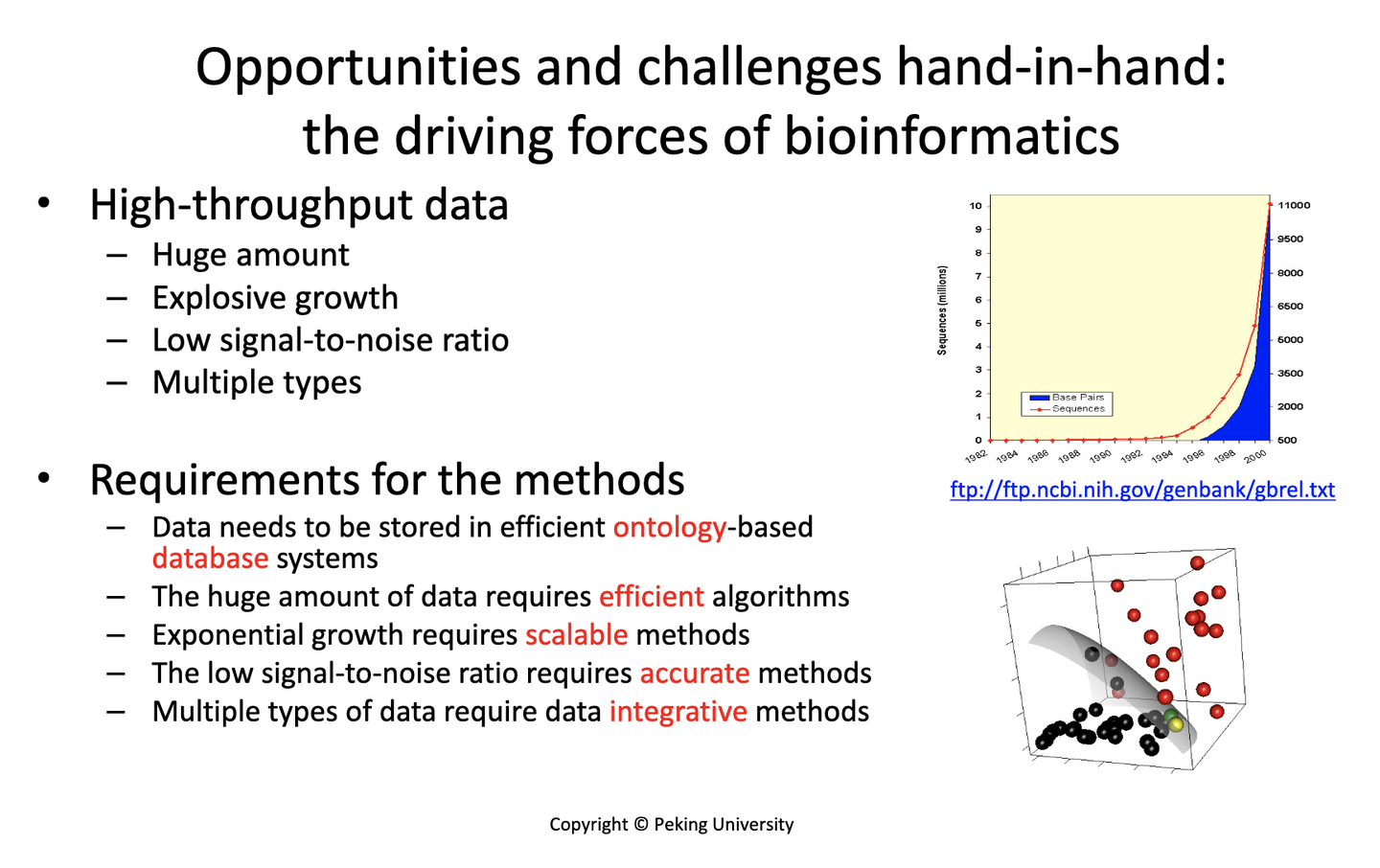 image-20200804235433871
image-20200804235433871
- 生物信息学这些年来的发展就是机遇与挑战并存的过程
- 生物信息学近年来非常快速地发展
- 生命科学和计算机科学的交叉应该是一个==必然的历史趋势==
- 生物信息学的定义到底是什么呢?
- 生物信息学是一个前沿交叉学科。
- 它主要通过开发并且应用计算和计算机技术来研究生物医学问题
- 主要的研究手段和开发的主要技术涉及计算机、数学、统计、和物理学里的技术。
 image-20200805000059142
image-20200805000059142
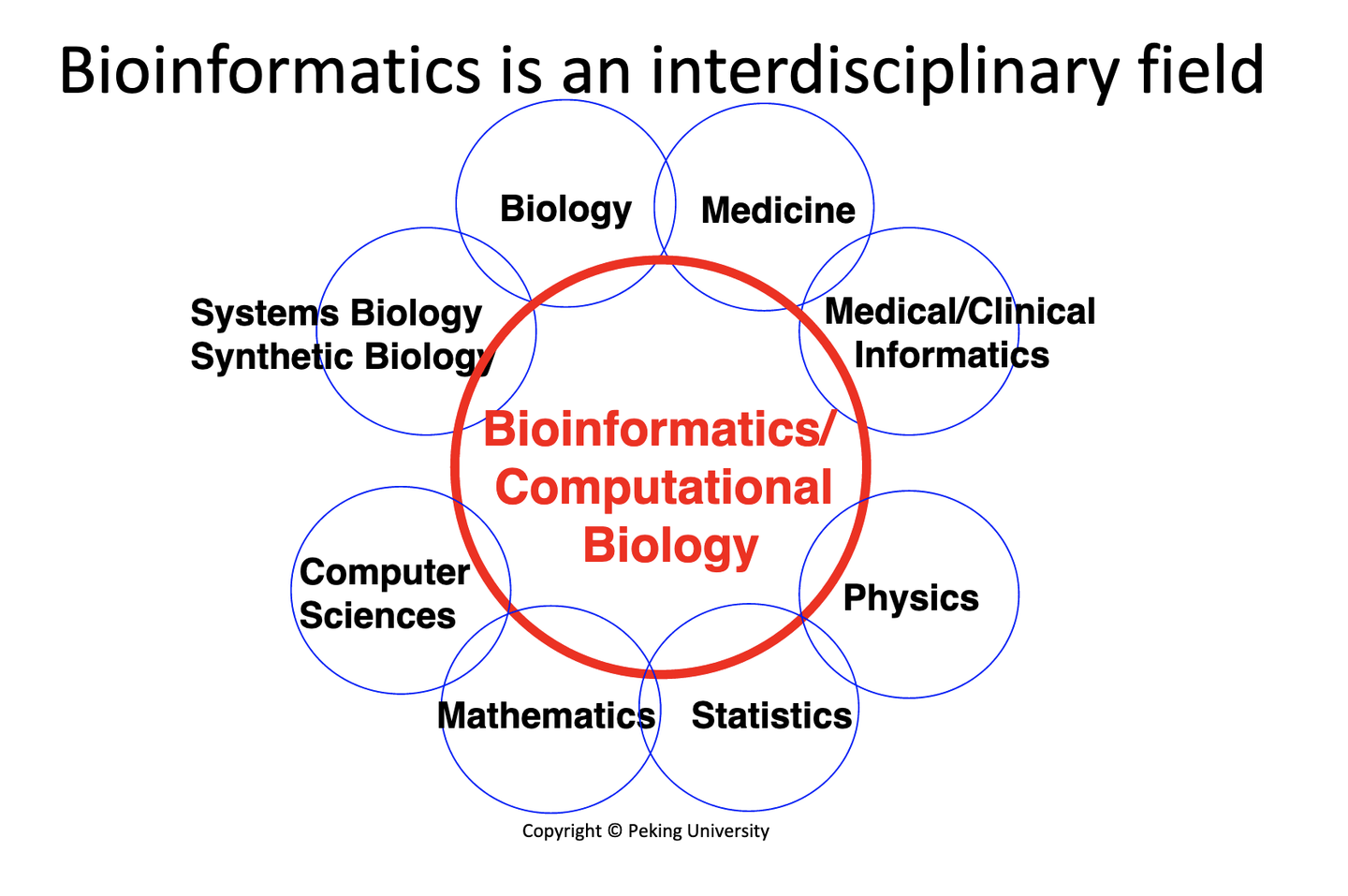 image-20200805000122582
image-20200805000122582
- 生物信息学有双重的身份
- 大家比较熟悉的是它作为一种技术:
- 它是一个管理、检索、和分析海量生物医学数据的==信息技术和计算技术==。
- 同时,生物信息学还是一种研究方法:
- 它是一种和传统生物学非常互补的一种==研究方法==。
- 它是一种自上而下,从全基因组出发,从系统水平出发, 基于数据的一种产生新假说、发现新规律、发现新功能元件的==研究方法==。
- 所以真正好的生命科学研究的趋势是把实验科学和生物信息学非常紧密的结合。
- 很多问题可以归纳在从基因型到表型围绕着中心法则这样一条主线。
- The Bio- in Bioinformatics:
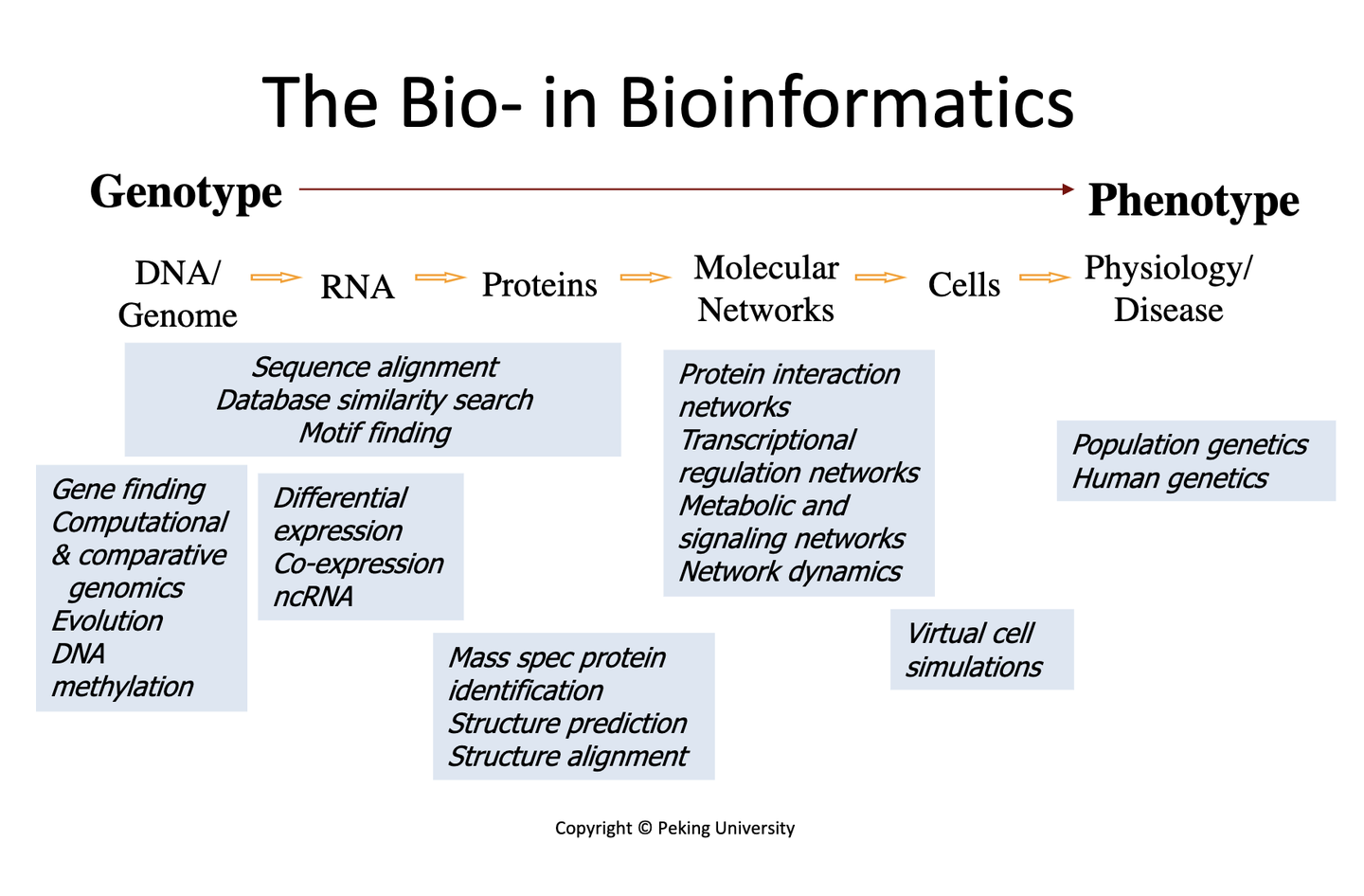 image-20200805000304255
image-20200805000304255
- The -informatics in Bioinformatics:
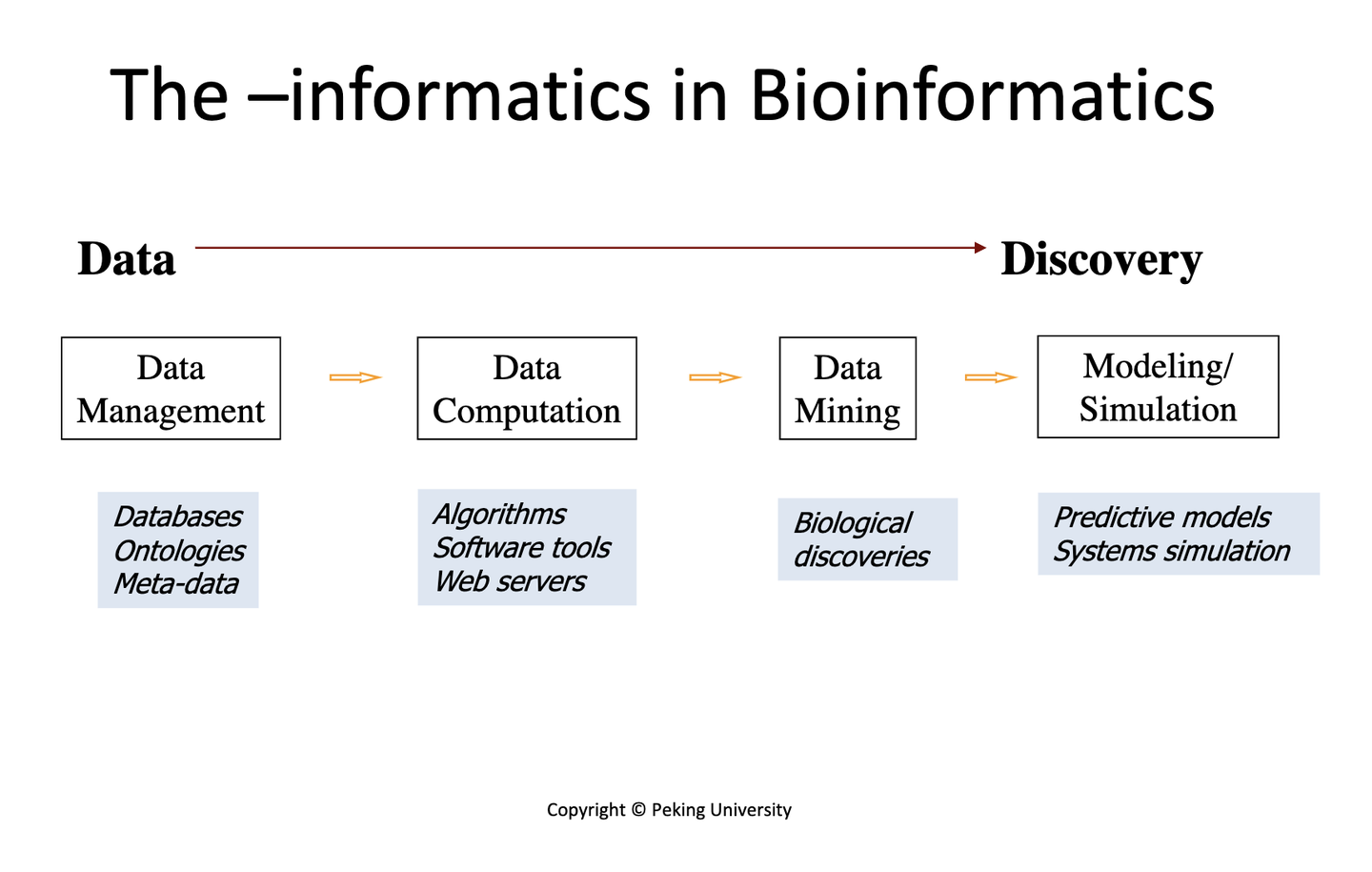 image-20200805000348407
image-20200805000348407
 image-20200805000607916
image-20200805000607916
- 我的回答:
- What is bioinformatics?
- 生物信息学是一个前沿交叉学科。
- 它主要通过开发并且应用计算和计算机技术来研究生物医学问题
- 主要的研究手段和开发的主要技术涉及计算机、数学、统计、和物理学里的技术。
- Can you name some of the biomedical questions studied in bioinformatics?
- The Bio- in Bioinformatics这张图中的topics
- Can you name some of the technical questions studied in bioinformatics?
- The -informatics in Bioinformatics这张图中的topics
- Why are you interested in bioinformatics?
- 21st century is the century of Biology!
 image-20200805001316916
image-20200805001316916
 image-20200805001610618
image-20200805001610618
- 抖个机灵~ 祝大家学习愉快~
1.3 生物信息学历史
 image-20200805125416273
image-20200805125416273
- 20世纪50 ~ 60年代:分子生物学的黄金时代
- 那个时候也是计算机领域的很多重要的概念和软硬件形成的阶段
- 20世纪70 ~ 80年代:分子生物学和计算机、互联网都逐渐成熟
- 20世纪90年代至今:分子生物学与计算机互联网飞速发展的一个阶段
- 1953年:Watson and Crick在Nature上报道了DNA的双螺旋结构,奠定了DNA作为遗传物质基础的地位。
- 20世纪50年代:
- 第一个蛋白的序列和第一个蛋白的结构被解析出来
- 1965年:Evolutionary divergence and convergence in proteins 发表
- Linus Pauling, Zuckerkandl
- 历史性的经典文献,分析haemoglobin蛋白,提出分子演化,==分子钟==概念
- 70页
- 1977年:Sanger DNA sequencing 发表。
- 一经发表了就一发不可收拾,因为它的效率比蛋白测序要高很多。
- 所以在七几年、八几年有大量的DNA序列开始被测量出来。
- 1988年:人类基因组计划启动。
- 美国能源部和NIH领导
- 有英国、法国、德国、日本和中国参与。
- 预算高达30亿美元。
- 它的主要目标除了测序人的基因组之外, 还包括对相关的模式动物的测序 以及对高通量技术的开发。
- 人类基因组计划和生物信息学关系是非常紧密的, 是一个相辅相成的关系。
- 人类基因组计划为生物信息学提供了大量的高通量技术所产生的数据
- 同时如果没有生物信息学,它这些数据也无法被分析。
 image-20200805125301658
image-20200805125301658
- Francis Collins(左起第一张照片),是人类基因组计划政府方负责人。
- Craig Venter(左起第二张照片),是当时产业界的负责人。
- James Watson(左起第四张照片) ,是人类基因组计划最早的倡导者、推动者和设计者之一。
- Maynard Olson(左起第五张照片), 也是美国科学院院士,是华盛顿大学教授。 人类基因组计划最早的一批非常重要的, 作出了重要贡献的人之一。 YAC和STS技术就是他主导发现的。
 image-20200805125520213
image-20200805125520213
- 1991年:诺贝尔奖获得者Walter Gilbert写了一篇短文, 说生命科学研究要开始经历一个==paradigm shift==. 在科学界paradigm shift是非常强的一个词, 是指范式的转换。 他说,在人的基因组被测出来之后, 生命科学的研究方法要有所转变了, 不再是只从一个基因出发,只看着周围一点地方, 而是说,研究都要从基因组水平出发 然后提出假设,再到实验室进行验证。
- 人类基因组计划在最开始早年是有很大争议的,受到很多人反对。 的确,在计划启动之后很久,基因组还没有被测序出来。
- 1995年:==细菌H. influenzae的基因组==被测出来
- Craig Venter用 shotgun sequencing ==鸟枪法==测出来的
- 1995年 ~ 2000年:大肠杆菌、酵母、线虫、果蝇的基因组都在下面的5年里被测序出来
- 2001年,人的基因组草图被发表。 当时政府和Craig Venter领导的Celera公司在同一天,一个在Nature上发了整整一期,一个在Science上整整发了一期。划时代的意义!
 image-20200805130006883
image-20200805130006883
 image-20200805130025000
image-20200805130025000
- 2004年:人的基因组完成图发表
- 2005年至今:从454开始,第二代测序技术发展起来,现在主要以Illumina HiSeq为主,NGS得到了越来越广泛的应用。
 image-20200805141746440
image-20200805141746440
- 生物信息学Bioinformatics这个词, 最早在文献里能找到这个词是在1970年。 一般大家,包括我本人,原来看到的是在1978年的时候 由Pauline Hogeweg发表的一篇文章里有Bioinformatics这个词。 但其实她和同事在1970年 在一个没有人知道的丹麦杂志里, 就定义了生物信息学的概念。 她当时定义的是,对生物系统的信息过程的研究。 这应该是生物信息学这个词最早出现的时候。 在生物信息学这个词出现之前, 就有一些领域的先驱做了一些现在看起来很简单,但是在当年是很概念性的很重要的工作。 我能找到的最早的满足我们现在的生物信息学定义的工作 是1951年在曼彻斯特第二届计算机大会上宣读的一篇文章, 后来在52上在Acta Crystallography杂志上发表。 这就是著名的结构生物学家John Kendrew 和写程序的Bennett合作的这篇文章, 用当时John Hopkins的EDSAC计算机, 来帮助解析蛋白结构,他们当时是在做肌红蛋白的结构。 主要是用计算机来做傅里叶变换。 这是51年的事情,最早的生物信息学的工作。
- 1977年, 专门存储蛋白三维结构的PDB(Protein Data Bank)数据库被建立。
- 同时在这个过程中, 大家认识到一个蛋白在自然界中会形成一个相对比较固定的结构。
- 有disordered region(无序区间)存在,但是有相对固定的结构。
- 20世纪70年代开始, 对蛋白的二级结构、三级结构的预测一直是生物信息学很重要的问题。 也是一个生物信息学的问题
- 比较著名的是74年的时候,Chou和Fasman的secondary structure prediction(二级结构预测)的方法。
- 后来,蛋白预测的方法越来越多,从94年开始到现在,一共已经有了20年的时间,[这期间]一直在举办一个叫CASP的竞赛——Critical Assessment of Structure Prediction,[该竞赛会]对各种蛋白结构预测方法进行比较。
- 1962年:COMPROTEIN。
- 除了结构之外,对序列的研究一直是生物信息学贯彻这么多年的一个重点。最早的工作是1962年的时候Margaret Dayhoff 写的一个计算机程序,叫做COMPROTEIN,它当时是在IBM7090,就比这个屋子还大的 这个巨大的计算机上,还不如你现在手里的一个小小的手机。
- 她这篇文章里所写的一些内容我觉得其实体现了为什么生物信息学是很重要的。比如她说,在1962年发表的时候,当时世界上已经有6个 蛋白的序列已经被知道了。那个时候每解一个蛋白就需要很多个生化专家坐在一起,因为他们需要把蛋白先 切成小的片段,然后再一个一个测出来再拼起来。这其实是一个组合(combinatorial)的问题, 在蛋白如果太长的时候,这就是很复杂的一个问题。所以,这个问题一定程度上也限制了 解出更多的蛋白。
- 所以当时Dayhoff就想,如果我们能够用计算机模拟专家做的这些事情,那今后普通的实验室都可以做了,不需要十几个专家坐在这里做这件事。
- 而且,有计算机来做,我们就 不只可以解6个蛋白,我们可以解60个蛋白。所以他们就做了这样的一个软件系统。 她除了做这个之外, 她还看到从62年开始有6个蛋白,过了几年居然就有了30个蛋白,已经增长的很快了,她就认识到,很多人已经开始没有办法继续跟踪这个领域的发展,所以她在65的时候做了第一个数据库,叫做 Atlas of Protein Structures and Sequences
- 1965年:Atlas of Protein Structures and Sequences**当时最开始的时候这就真的是印出来的一本书,65年时就包括了世界上所有65个蛋白序列。 这个Atlas后来就演变成网上的电子数据库,就是83年的Protein Information Resource,PIR,之后被UniProt取代。
- 1982年:GenBank被建立
- Dayhoff她做了很多很重要的工作。她本科是学数学的。Ph. D. 是学化学的。当时她做的工作都是在 National Biomedical Research Foundation,在Maryland的一个机构做的。她除了做了这些计算机算法和数据库, 她也意识到,有了这些同一家族的若干个蛋白之后,可以来构建一个演化的历史。 所以她在66年的时候, 在Science上发表了这篇文章,对当时已知的Ferredoxin的序列进行了分析,研究了Ferredoxin这个家族的演化。
- 在同一个阶段,另外一个演化上非常重要的生物信息学的工作,是在1967年由 Walter Fitch 和 Emanuel Margoliash 发表的这样一篇文章。在这篇文章里,他们第一次发表了这样一个方法、同时配备了一个计算机程序做系统发育树的构建。 后来,我准备课程时,挖掘出来了Fitch在84年写的一个小回路的一个东西。他当时在66年的时候, 就认识到,这个家族已经有了 10个蛋白质,所以他当时就意识到,未来通过这些蛋白做演化的研究、系统发育树的构建会是一个很重要的工作。 所以他就写了一个计算机程序来做这样一个事情。那个时候,他在 Wisconsin 大学做助理教授 所以他66年就在吭哧吭哧自己做这个事情,当时他选择的研究对象就是Cytochrome C(细胞色素C),因为Cytochrome C这个家族在那个时候有10个蛋白, 在不同物种中是被知道的。这是已知蛋白序列最多的一个家族。然后在66年的时候, Emanuel Margoliash 到 Wisconsin 大学去访问,讲了他做的Cytochrome C的一些工作, 提到说他实验室还有10个Cytochrome C蛋白序列还没有发表。于是,他们两个就合作, 利用这20个蛋白——这就是当年的海量的数据——利用 Walter Fitch 的计算机程序。否则的话,20个序列你如果真的构建系统发育树,其实在人工上就已经是很难很难的了, 不是特别可能了。因为有他的计算机程序,他们就作了这样一个工作,这也是 非常经典的、在演化生物学和生物信息学都非常经典的一个很重要的工作。
- 再往后发展,知道的蛋白越来越多,那这时就经常需要把两个蛋白拿来做匹配。最开始大家做的时候, 就是测了一个 human的 protein ,再测个 mouse,其实相似度很高,那你做 sequence alignment其实是很简单的,肉眼就可以做。 但如果两个物种离的比较远的时候,它们的序列的相似度低很多, 你如何找到这两个序列的最佳匹配呢?什么样的 alignment 是最佳的alignment呢?其实这在计算上是一个计算量很大的问题。
当时已经成为了一个瓶颈的问题。 - 1970年:Needleman-Wunsch global alignment(全局比对)。所幸在1970年, Saul Needleman 和 Christian Wunsch 就在 Journal of Molecular Biology 杂志上发表了一篇文章报导了一个算法,可以用计算机很快地来找到两个蛋白匹配的最优解,它用的是 dynamic programming (动态规划)的一个研究思路。 ==这个工作也是一个历史性的非常重要的工作==。当时大家就非常happy(幸福)地用了若干年。 但是到70年代末的时候,作序列比对的时候,你把这样两个序列比对起来,其中很重要的一个步骤是,对在一起的两个氨基酸 是合适的还是不合适的。比如,valine(缬氨酸)和 leucine(亮氨酸)如果发生了替代,其实对于结构和功能不会有太大影响。如果一个 Cystein(半胱氨酸) 被一个 Proline (脯氨酸)替代, 那这个其实会对结构和功能有极大的影响。所以如果真正这两个同源的话,这个 valine (缬氨酸),这个 cystein(半胱氨酸)和proline(脯氨酸) 被align(被比对在一起)的概率其实应该是很小的。那怎样对这个有一个量的评估?这就又还是Margaret Dayhoff。
- 1978年:Margaret Dayhoff发明了 PAM matrix,用来评估amino acid substitution
- 她根据演化的一些信息,从65年就开始收集蛋白序列,然后通过她的生物学知识知道哪些序列大概是一个家族,然后找同源性比较高的 手工地做 alignment,然后就开始数,在每一个位置,不同物种之间,有什么样的“氨基酸到哪个氨基酸的替换”,发生的频率有多少,然后根据这样的一个信息,她就构建了一个 substitution matrix (替换矩阵)。 这个 substitution matrix (替换矩阵)从78年开始,一直就是生命科学领域的最权威的一个 substitution matrix (替换矩阵),一直到92年的时候,被 BLOSUM matrix取代。
- 1992年:一直到92年的时候,被 BLOSUM matrix取代
- 现在多数的方法是用 BLOSUM。但在七几年的时候,当时生物学发现基因是有 intron(内含子) 的, 这样你就可以想像,Needleman-Wunsch它是一个 global alignment(全局比对),它把两个序列从头到尾都 align起来。 但基因有 intron (内含子)的话……这个 intron (内含子)突变率很高(其实是无所谓的)主要还是 exon (外显子)的 alignment的 quality是更为重要的。那你如何处理这样的一个事情呢?就是==一个序列里有特重要的区间、有特不重要的区间,你怎么办==?==这就是 Needleman-Wunsch global alignment(全局比对)无法处理的一个问题。==
- 当时 Temple Smith 和 Michael Waterman 他们,Waterman自己会讲的更好的。我之前问过他,他就说,其实他们两个当时是花了两三年时间在提高 Needleman-Wunsch 的 algorithm(算法),结果突然就听说 基因有 intron(内含子),自己做两三年的工作发现并不那么重要,这个新的问题怎么解决呢?
- 1981年:Smith-Waterman alignment(局部比对)。
- 所以他们在81年的时候发表了这篇,也是在 Journal of Molecular Biology 发表的一篇文章,能够找到两个序列之间的最优的局部片段的比对。所以,如果两个基因,你不管它intron(内含子)差的多多, 它能够把相似度最高的两个 exon (外显子)给你找出来、比对起来。
- 所以这就非常好地解决了刚才说的一个序列里有重要、有不重要区间的问题。 他们的这个方法非常非常的巧妙,这个高歌老师他会讲。其实就是在前面的 Needleman-Wunsch 的算法里有那么两个非常小的改动,但是就非常巧妙地解决了这个问题。
- 然后在八几年,蛋白和基因的序列越来越多,这时大家经常需要做的一个事情是:我所研究的这个蛋白,是不是 和某一个已知的蛋白序列上有相似性呢?如果有的话,我是不是可以利用已知蛋白的功能来指导我的实验呢?最开始,大家就是用 Smith-Waterman 方法来做, 但是在序列太多了之后,Smith-Waterman算法都已经太慢了,这个时候就又进入了一个瓶颈的阶段,所幸就是在90年的时候, Stephen Altschul 和 David Lipman 等人发表了 BLAST 方法。
- 1990年:Stephen Altschul 和 David Lipman 等人发表了 BLAST 方法
 image-20200805144910875
image-20200805144910875
- 1997年: Gapped BLAST & PSI-BLAST
- 之后他们又在 97年的时候发表了一个允许有插入的 Gapped BLAST
- 和运用多序列比对的 PSI-BLAST 方法。
- 就解决了在一个序列数据库里找到和你感兴趣的基因或蛋白最相似的 序列的这样的一个方法。
- ==BLAST 算法的这两篇文章,90年和97年,按照 Google Scholar 的统计,都已经引用了将近5万次。 每一篇文章被引用了将近5万次,这是在整个科学领域里都是排在前十名的,所以这是非常重要的 一个工作。==
- 20世纪90年代:以 EST 和芯片技术为代表的研究基因表达的高通量技术发展的非常快,所以当时就有了一系列的对芯片基因表达的研究。
- 同时,大家可能记得95年细菌基因组开始被测序出来之后,很多物种的全基因组开始被测序出来,于是就有了大量的从一个基因组里预测基因。
- 1997年:Christ预测方法和Genscan预测方法。
- 做基因 prediction(预测)比较有代表性的就是 1997年 Christ(全称Christopher) Burge, 和 Samuel Karlin 发表的 Genscan 的这一个预测方法。
- 并且,只要有了两个 Genome (基因组),马上你就要想这==两个 Genome (基因组)的相似度有多少呢?最相似的是什么呢?==
- 所以这就是要做 一个全基因组比对,所以就有一系列全基因组比对的方法。 然后你就还想知道,我感兴趣的、正在研究的这个基因,它是在基因组的哪一个位置呢?它的上下游有什么可能的调控区域呢?
- 2002年:BLAT算法。UC Santa Cruz 的 Jim Kent 发表了 Blat 算法
- 它能够==把一个 感兴趣的序列很快找到它在基因组上的位置==【很有用!】。
- 之后他又发表了一个方法,能够把两个基因组作比对。
- 2005年开始,新一代测序技术被发明出来以后,数据量越来越多地在积累。于是
- 2007年:SRA数据库成立。
- 我前面提到的专门存储新一代测序技术的SRA数据库被建立起来。
- 也是从05年之后,生物信息学越来越多的研究是围绕着新一代测序的数据分析, 因为这些还是有最大的数据量。
- 一个崭新的一个技术,有可能给你带来最多的科学发现。
- 前面,大家应该可以想像,生物信息学它对生命科学的贡献其实还是比较显而易见的。
- 其实它对计算机科学也有很多的贡献,完全并不是说只是使用计算机开发好的一些技术。比如,==神经网络,遗传编程等,其实都是从生命科学的现象出发,倒推回一个 崭新的一个算法之后,被应用到了其它的很多的领域==。
- 另外一个例子是我(魏老师)当时在 Stanford (斯坦福)读 Ph. D. 的时候的一个师弟,他当时就学完 BLAST 方法之后, 他就认识到,这个方法呢,可以用在当时的互联网 routing (路由)的问题上。 他有一个弟弟在 Stanford computer science系读 Ph. D. , 于是他们两个就真的根据 BLAST 写了一个计算机程序,来做 Internet的routing (路由), 结果就成立了一个公司, 2000年成立了一个公司,到04的时候就被 Jupiter 以三亿三千七百万美元收购。 所以, 他真的就是从一个 BLAST 方法受到的启发,完全应用到互联网领域。
- 所以这种跨界的应用有时候是非常非常有效的。在早年的时候,就像当时我(魏老师)们在念生物信息学的时候, 全世界就没有几个生物信息学的专业。这个是03年别人写的一篇综述里统计的,早年有生物信息学专业的十个地方,包括 Stanford和 Boston (波士顿), University of London (伦敦大学)。现在完全发表生物信息学文章的杂志,这里就已经有列出来了19个。
- 另外还有像 Nucleic Acids Research , Genome Research , Nature Methods , Nature Biotech这种,其实它不是生物信息学杂志,但是它发表大量的 生物信息学相关的文章,所以现在这个领域已经比当年我们念书的时候要舒服。这些年它发展是很快的, 当时我们统计了一下,你如果看 PubMed 里所有的生命科学文献,从97年到现在这15年增长的趋势, 你们可以看到,一直是增长的。那如果你要看 PubMed 里生物信息学文献的增长趋势,是要陡峭很多的。 你如果要算一下,每一个时间点,生物信息学文章占整个生命科学文章的百分比的话,你会看到这个百分比都在增加,就是说==生物信息学在整个生命科学 领域里的分量越来越重==。那到12年的时候,就大概有1.6%多的 生命科学文章是生物信息学文章。那你可能会想到,在中国的情况会是什么样子的。 所以下面一节,我就给大家介绍一下中国的生物信息学。
 image-20200805150943919
image-20200805150943919
- Current Bioinformatics Journals:
- Bioinformatics
- BMC Bioinformatics
- BMC Systems Biology
- Briefings in Bioinformatics
- Bulletin of Mathematical Biology
- Cancer Informatics
- Computational Biology and Chemistry
- Computers in Biology and Medicine
- Database: The Journal of Biological Databases and Curation
- IEEE/ACM Transactions on Computational Biology and Bioinformatics
- Journal of Bioinformatics and Computational Biology
- Journal of Biomedical Informatics
- Journal of Computational Biology
- Journal of Integrative Bioinformatics
- Journal of Mathematical Biology
- Journal of Theoretical Biology
- PLOS Computational Biology
- Source Code for Biology and Medicine
- Statistical Applications in Genetics and Molecular Biology
- Current Journals which is interested in Bioinformatics.
- ==Nucleic Acids Research==
- ==Genome Research==
- ==Nature Methods==
- ==Nature Biotechnology==
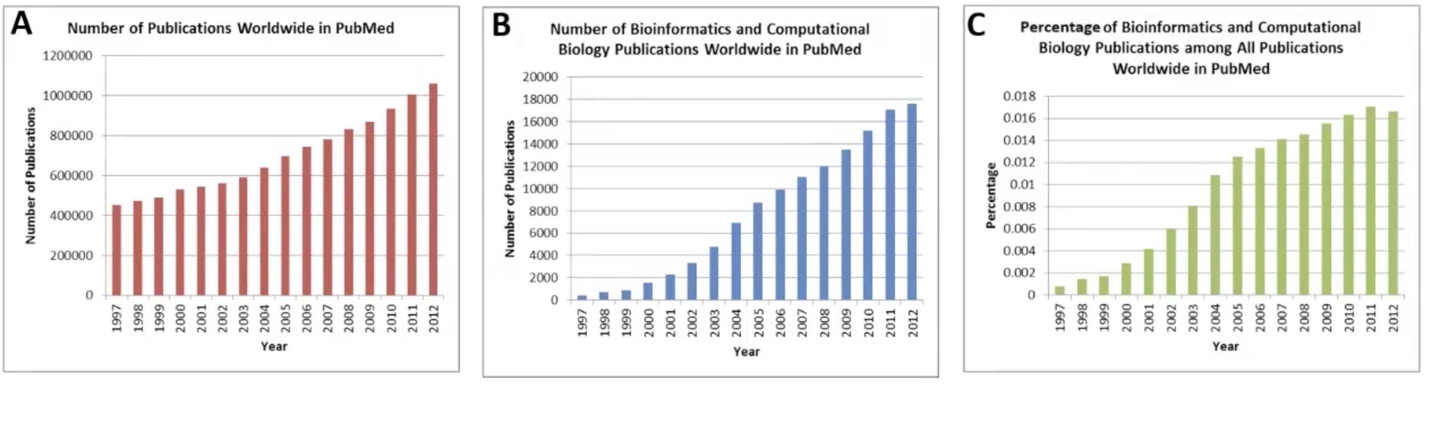 image-20200805151744904
image-20200805151744904
 image-20200805151835128
image-20200805151835128
- 我的回答:
- What are the main events in biology that are critical for the development of bioinformatics?
- 上面标志性事件中关于Biology的
- What are the main events in computer sciences?
- 上面标志性事件中关于Computer Science的
- Can you name a few of the seminal works on the analysis of sequences and structures?
- 如全局比对算法、局部比对算法、BLAST、BLAT、PAM矩阵
- Can you name any of the early pioneers?
- 去上面找人名儿就行
- Are there any important works and/or pioneers that are missed in this presentation?
- 布吉岛~,等以后资历深了再回来答这题
1.4 中国大陆的生物信息学
 image-20200806133736833
image-20200806133736833
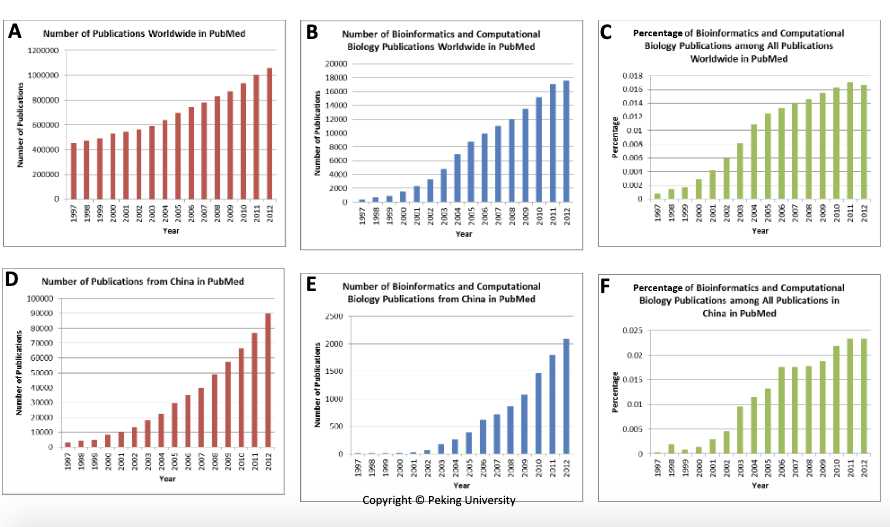 image-20200806133902541
image-20200806133902541
- 2012年中国生物信息学文献占中国生命科学总文献的比例为2.4%, 而国际上为1.7%
- 因此,虽然中国的生物信息学起步比较晚, 但是自上世纪90年代以来, 发展的势头是非常的好的。
- 中国生物信息学发展的驱动力主要有四点。
- 第一,是计算机技术和互联网的发展。 1994年,中国和世界的 TCP/IP 互联网连接首次建立, 就是北京中科院高能物理所和斯坦福大学线性加速器之间的拨号连接。 紧接着,1994年5月17号, 与 FIX-West 的官方连接建立, 美国能源科学网络 ESnet 同意开始接受中国的IP流量。 之后,中国互联网开始了飞速的发展。 根据中国互联网信息中心统计,到2012年底,中国互联网用户已经达到五亿六千四百万, 其中城市居民互联网使用率达到59.1%,农村23.7%。
- 中国生物信息学发展的第二个推动力,是国内外基因组学的发展。 1998年,中国国家人类基因组南方中心与北方中心,分别在上海和北京成立。==1999年,华大基因中心在北京成立,==后迁往深圳, 是目前国际上最大的基因组测序中心之一。 ==2003年,中科院北京基因组所成立==, 中国完成了人的基因组的1%的测序,以及水稻和之后很多物种的全基因组测序。 这给生物信息学提供了大量的珍贵的数据。
- 中国生物信息学发展的第三个推动力,是自上世纪80年代末开始的,政府科研经费越来越多的投入, 主要的科研经费来源于科技部和自然科学基金委。 科技部于1986年3月启动“863”计划,支持科学技术研究。 于1997年3月启动“973”计划,支持大型基础科学研究。 之后又启动了蛋白质研究计划,重大科学问题等一系列大规模的研究支持计划。 自然科学基金委员会支持科学研究项目, 同时也有国家杰出青年,国家优秀青年等人才支持计划。
- 中国生物信息学发展的第四个推动力,是越来越多地投身于这个领域的本土和海外引进的人才以及本土培养的学生。 最早的从上世纪80年代末90年代初开始, 国内一些来自物理、化学、数学、自动化, 和生物学领域的先驱,就在当时非常紧张的经费条件下,开始进行了生物信息学研究。 研究方向包括: 系统发育树构建,非编码RNA,DNA序列分析,表达调控,结构预测与分析,药物设计,分子网络, 基因组演化,等等。 我(魏老师)比较熟悉的例子包括:
- 郝柏林,陈润生,张春霆,李衍达,施蕴渝,罗辽复,丁达夫,孙之荣,来鲁华,顾孝诚,罗静初等教授。
- 很遗憾的是顾孝诚教授于2012年11月在北京去世。 ==顾孝诚和罗静初教授于1997年 在北京大学成立了中国第一家生物信息学中心==。
 image-20200806134312628
image-20200806134312628
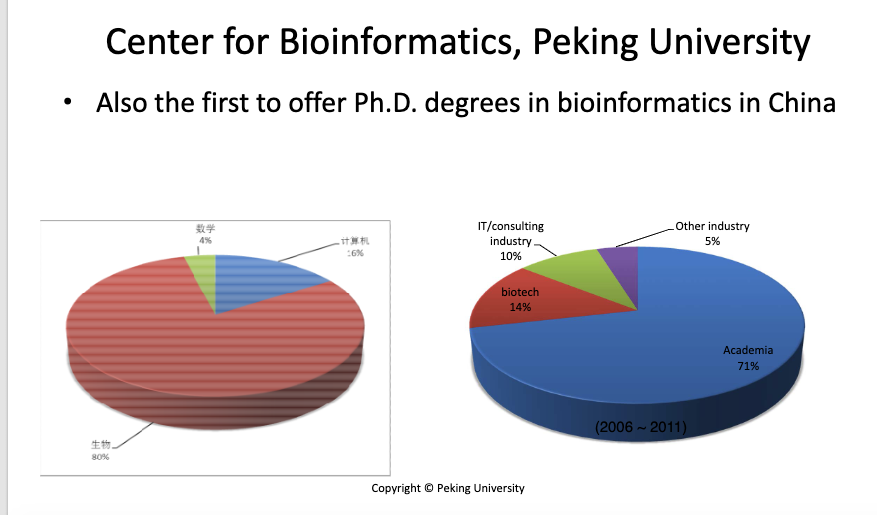 image-20200806134338058
image-20200806134338058
- 北京大学CBI中心的研究工作将计算和实验相结合, 一直受到科技部,基金委,和教育部的多方支持。 在2000年,CBI中心在中国率先授予生物信息学博士学位。 至今培养了一大批优秀的博士毕业生。 学生的来源80%有生物学背景,20%有计算机、数学和其它的背景。 毕业生里,71%至今仍留在科研院校做研究工作,其中很多已经是教授、博导;14%在生物技术公司工作; 15%在IT和其他产业工作。
 image-20200806134409876
image-20200806134409876
 image-20200806134422399
image-20200806134422399
 image-20200806135558397
image-20200806135558397
1.5 阅读参考
关于PubMed的使用
点击了PMID链接后,你会看到一个新页面,这个页面上描述了这篇文章的摘要。若需获取全文的链接,可以点击该页面右上角、在“Search”按钮下方的全文链接,链接以图片显示。该图片上面应该有些像“Free final text”,“Free in PMC”之类的字样。图片本身具体的样式依赖于这篇文章所发表的期刊(有时还要取决于文章本身)。
必读文章
- Bioinformatics. 2003 Nov 22;19(17):2176-90. Early bioinformatics: the birth of a discipline--a personal view. Ouzounis CA, Valencia A. PMID: 14630646
- PLoS Comput Biol. 2011 Mar;7(3):e1002021. doi: 10.1371/journal.pcbi.1002021. Epub 2011 Mar 31. The roots of bioinformatics in theoretical biology. Hogeweg P. PMID: 21483479
- PLoS Comput Biol. 2008 Apr 25;4(4):e1000020. doi: 10.1371/journal.pcbi.1000020. Bioinformatics in China: a personal perspective. Wei L, Yu J. PMID: 18437216
补充阅读
- Nature. 1991 Jan; 349(10):99 Towards a paradigm shift in biology. Gillbert W.
1.6 测试题
 image-20200806134920203
image-20200806134920203
1.7 参考答案
尽量独立做题,回来再看看答案~
bioinfo-PKU_bioinfo_week1 ~ Herman's Blogzhaohuanan.cc